DNA鑑定

DNAの基本
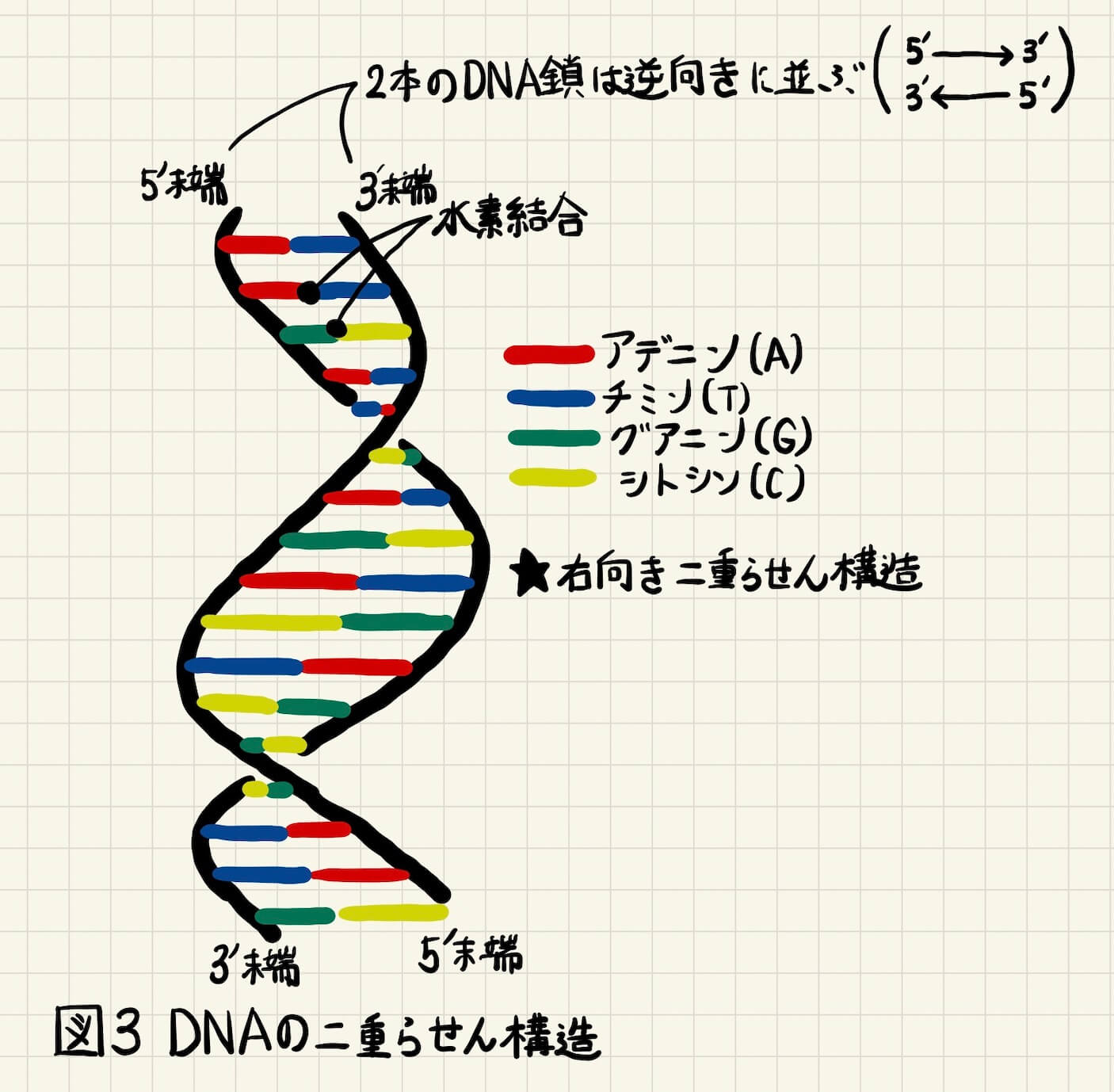
DNA(デオキシリボ核酸)は、遺伝情報を保持する分子であり、主に細胞核内に存在する。
DNAは以下から構成される:
• 糖(デオキシリボース)
• リン酸
• 塩基(A, T, G, C)
塩基は、A–T、G–C の組み合わせで結合し、二重らせん構造を形成する。
法科学では、個人差の大きい STR(短鎖反復配列) が主に解析対象となる。
PCR法(Polymerase Chain Reaction)
PCR法とは、DNA分子の特定の領域を繰り返しコピーし、指数関数的に増幅させる技術。
PCRの3つの基本ステップ
通常、これらのステップを25〜35サイクル繰り返す。
1. 熱変性 (Denaturation): 高温(92°C〜95°C)で2本鎖DNAを単鎖に分離する 。
2. アニーリング (Annealing): 温度を下げ(50°C〜65°C)、プライマーを標的領域の末端に結合させる 。
3. 伸長 (Extension): 中間の温度(通常72°C)で、DNAポリメラーゼが新しいDNA鎖を合成する 。
- アデニル化(A-tailing): 伸長ステップの最後に、3'末端に余分な塩基(A)を付加することで、増幅産物のサイズを一定にする 。
※実習で使ったプログラムは、initial denaturation 4min 94°C, (1. Denatuation - 1min 94°C, 2. Annealing - 1 min 57°C, 3 Elongation – 1min 72°C) ×30 cycles, final elongation 4min 72°C, then 0°C, 冷凍保存後、ゲル流動(gel electrophoresis)で乖離させる
主要な構成要素
• 鋳型DNA (Template DNA): 増幅の対象となるDNA。市販キットでは通常0.5〜2.5ngが必要。
• プライマー (Primers): 特定の領域を定義する短い合成DNA。
• DNAポリメラーゼ: 新しいDNA鎖の合成を触媒。耐熱性のTaqポリメラーゼが広く使われている。
• dNTPs (deoxynucleoside triphosphates): DNAの構築ブロック(A, T, C, G)
• バッファーと塩: pHの維持や、ポリメラーゼの働きを助けるマグネシウムイオンを含む
分析上の注意点
• スタッター (Stutter): DNA合成中の滑りにより、真のアレルより1反復分短いピークが出現する現象
• 不完全なアデニル化: 3'末端へのA付加が不完全だと、ピークが2つに分かれて見える(スプリットピーク)原因になる
• プライマー・ダイマー: プライマー同士が結合して増幅されてしまい、反応効率が低下することがある
• 阻害物質 (Inhibitors): 血液中のヘマチンなどが原因で、増幅が阻害され不完全なプロファイルになることがある
DNA鑑定結果の評価
PCRの鑑定結果は主に尤度比(likelihood ratio)を用いて評価される。
ハーディー・ワインベルグの法則 (HWE)
• 定義: 無作為に交配する集団において、各遺伝子座のジェノタイプ頻度は世代を超えて一定に保たれるという原理。(p2+2pq+q2=1)
• HWEが成立するための条件: 無限に大きな集団、ランダムな交配、移住がないこと、自然選択がないこと、突然変異がないこと
• 偏差の原因: 実際の人間集団では、集団の有限性(遺伝的浮動)、非ランダムな交配、移住(ワールンド効果)、自然選択、突然変異などによりHWEから偏差が生じることがある。
頻度の計算(Allele, Genotype, Profile)
• アレル頻度(Allele Frequency): 適切なアレルデータベース(少なくとも500人以上のサンプルが推奨)を用いて算出する
• 計算式:特定のバレルの出現数 ÷ データベース内の全アレル数
• ジェノタイプ頻度(Genotype Frequency): ハーディー・ワインベルグの法則(HWE)に基づき算出する (p2+2pq+q2=1)
― ホモ接合体(Homozygotes): p2 or q2
― ヘテロ接合体(Heterozygotes): 2pq
• プロファイル頻度(Profile Frequency): 各遺伝子座(Locus)のジェノタイプ頻度を積の法則で掛け合わせて算出する。
統計的補正と法医学的ガイドライン (FSR Guidance)
• 稀なアレルの補正: データベースに存在しないアレルが発見された場合、最小アレル頻度(例:0.01や5/2N)を設定して調整する。
• サンプリング・バイアスの補正: データベースのサイズが小さい場合の不確実性を考慮し、Baldingらの手法(証拠サンプルと参照サンプルをデータベースに加えて再計算する等)を用いる。
• 亜集団(Subpopulations)の補正: 近縁関係による一致確率の上昇を考慮するため、シータ値(通常0.03〜0.05)を用いた補正を行う。
• 上限原則(Ceiling Principle): 英国などでは、一致確率の最小値を「10億分の1」として報告する慣例がある。
特殊な解析
ハプロタイプ(Haplotype): ミトコンドリアDNAやY染色体は単一の遺伝子座として遺伝するため、メンデルの法則に従わない 。頻度は「観察数 / 全体数」で計算される 。
親子鑑定(Paternity Testing):
• Paternity Index (PI): 「その男性が父親である確率」と「無関係な別の男性が父親である確率」の比で評価する
• Combined Paternity Index (CPI): 複数の遺伝子座のPIを掛け合わせたもの
Y-STR分析
Y-STRとは、Y染色体上に存在する短鎖反復配列を解析する方法である。
Y染色体は父系遺伝するため、同じ父系男性は同一または極めて類似したY-STRプロファイルを持つ。
Y-STRの主な特徴と構造
• NRY(Y染色体非組み換え領域): Y染色体の約95%を占め、ここにあるマーカーが分析対象となる。
• ハプロタイプ(Haplotype): Y染色体上のマーカーはセットで遺伝するため、個々の遺伝子座としてではなく、一つの「ハプロタイプ」として扱われる。
―> このため、頻度計算に「積の法則」は使えず、数え上げ法(Counting Method)が用いられる
Y-STR法の主な用途
• 混合試料の分析(性犯罪など)
大量の女性DNA(被害者)の中に微量の男性DNA(加害者)が含まれる場合、常染色体分析では男性側のピークが埋もれてしまいますが、Y-STRなら男性成分のみを特異的に検出できます。
• 父系血縁関係の確認
遺体識別や、数世代前の父系祖先の特定に利用されます。
• 行方不明者捜査
父親や兄弟などの親族から参照サンプルを採取して照合が可能です。
実習で行ったPCR法の手順
1. 滅菌スワブで採取した頬細胞を1mLのバッファー溶液に入れ、遠心分離によって細胞をペレットとして沈殿させた後、不要な上澄み液を除去する
2. 細胞のペレットに、金属イオンを吸着してDNA分解酵素(DNase)の働きを阻害する10% Chelex樹脂を250μL加え、均一になるまで激しく攪拌する
3. 混合液を65℃で20分間、続いて100℃で5分間加熱し、熱によって細胞膜を破壊してDNAを液中に放出させる
4. 再び遠心分離を行い、Chelex樹脂や細胞の残渣を沈殿させ、DNAが含まれる上澄み液をテンプレートDNAとして回収する
5. 滅菌チューブに、DreamTaq Master Mix(耐熱性DNAポリメラーゼ、dNTPs、マグネシウムイオンを含むバッファー、グリセロール、追跡用染料を混合したもの)を10μL分注する
6. さらに、標的領域を挟み込むためのプライマーペア5μL、分子生物学グレードの滅菌水3μL、および抽出したテンプレートDNA 2μLを加え、合計20μLの反応液を調製する
7. サーマルサイクラーを用い、まず94℃で4分間の初期熱変性を行い、その後、熱変性(94℃・1分)、アニーリング(57℃・1分)、伸長(72℃・1分)のサイクルを30回繰り返すことで、特定のDNA断片を指数関数的に増幅させる
8. アガロース粉末2.25gを150mLのTBEバッファー(Tris-Borate-EDTA)に加熱溶解させ、DNAを可視化するための蛍光染料であるMidori Greenを5μL添加してゲルを作成する
9. 電気泳動槽にランニングバッファーを満たし、ゲルのウェル(溝)に増幅したPCR産物20μLと、サイズ比較用のDNAサイズマーカー5μLをロードする
10. 100Vの電圧で約1時間電気泳動を行い、負に帯電したDNA断片を正極方向へ移動させる
11. UVイルミネーターで紫外線を照射し、Midori Greenと結合したDNAの発光を撮影することで、増幅されたDNA断片の有無とサイズを確認し、個人の識別を行う
R
